2.5.4
DBSCAN
- DBSCAN agrupa puntos en función de la densidad local, lo que permite detectar formas arbitrarias y dejar las regiones poco densas como ruido.
- Dos hiperparámetros dirigen el algoritmo:
eps(radio del vecindario) ymin_samples(número mínimo de vecinos para declarar un punto como núcleo). - Cada muestra se etiqueta como núcleo, borde o ruido; los clústeres son componentes conectadas de puntos núcleo más los bordes que los tocan.
- Una receta habitual es fijar
min_samples(≥ dimensión + 1) y barrerepsmientras se observa el porcentaje de puntos que pasan a ruido.
Intuicion #
Este metodo se entiende mejor al conectar sus supuestos con la estructura de los datos y su efecto en la generalizacion.
Explicacion Detallada #
1. Descripción general #
DBSCAN no necesita conocer cuántos clústeres habrá. Analiza cada punto:
- Puntos núcleo: tienen al menos
min_samplesvecinos a una distancia ≤eps. - Puntos borde: se encuentran dentro del radio
epsde algún núcleo pero no cumplen el criterio por sí mismos. - Ruido: no pertenece al vecindario de ningún núcleo.
Gracias a esta visión basada en densidad, DBSCAN maneja clústeres no convexos (dos lunas, anillos, etc.) y descarta automáticamente zonas dispersas. Escala siempre las variables antes de ajustar.
2. Definición formal #
Para (x_i \in \mathcal{X}), su vecindario (\varepsilon) es
$$ \mathcal{N}_\varepsilon(x_i) = \{ x_j \in \mathcal{X} \mid \lVert x_i - x_j \rVert \le \varepsilon \}. $$Si (|\mathcal{N}_\varepsilon(x_i)| \ge \texttt{min_samples}|), el punto es núcleo. DBSCAN expande clústeres recorriendo puntos alcanzables por densidad y marca como ruido los no visitados. Con un índice espacial la complejidad es (O(n \log n)).
3. Ejemplo en Python #
El siguiente script usa scikit-learn para ejecutar DBSCAN sobre un conjunto de dos lunas con ruido, colorea puntos núcleo/borde y reporta cuántos quedan como ruido.
from __future__ import annotations
import japanize_matplotlib
import matplotlib.pyplot as plt
import numpy as np
from numpy.typing import NDArray
from sklearn.cluster import DBSCAN
from sklearn.datasets import make_moons
from sklearn.preprocessing import StandardScaler
def run_dbscan_demo(
n_samples: int = 600,
noise: float = 0.08,
eps: float = 0.3,
min_samples: int = 10,
random_state: int = 0,
) -> dict[str, int]:
japanize_matplotlib.japanize()
features, _ = make_moons(
n_samples=n_samples,
noise=noise,
random_state=random_state,
)
features = StandardScaler().fit_transform(features)
model = DBSCAN(eps=eps, min_samples=min_samples)
labels = model.fit_predict(features)
unique_labels = sorted(np.unique(labels))
cluster_ids = [label for label in unique_labels if label != -1]
noise_count = int(np.sum(labels == -1))
core_mask = np.zeros(labels.shape[0], dtype=bool)
if hasattr(model, "core_sample_indices_"):
core_mask[model.core_sample_indices_] = True
fig, ax = plt.subplots(figsize=(6.2, 5.2))
palette = plt.cm.get_cmap("tab10", max(len(cluster_ids), 1))
for order, cluster_id in enumerate(cluster_ids):
mask = labels == cluster_id
color = palette(order)
ax.scatter(
features[mask & core_mask, 0],
features[mask & core_mask, 1],
c=[color],
s=36,
edgecolor="white",
linewidth=0.2,
label=f"Cluster {cluster_id} núcleo",
)
ax.scatter(
features[mask & ~core_mask, 0],
features[mask & ~core_mask, 1],
c=[color],
s=24,
edgecolor="white",
linewidth=0.2,
marker="o",
label=f"Cluster {cluster_id} borde",
)
if noise_count:
noise_mask = labels == -1
ax.scatter(
features[noise_mask, 0],
features[noise_mask, 1],
c="#9ca3af",
marker="x",
s=28,
linewidth=0.8,
label="Ruido",
)
ax.set_title("Demostración de DBSCAN")
ax.set_xlabel("Característica 1")
ax.set_ylabel("Característica 2")
ax.grid(alpha=0.2)
ax.legend(loc="upper right", fontsize=9)
fig.tight_layout()
plt.show()
return {"n_clusters": len(cluster_ids), "n_noise": noise_count}
resultado = run_dbscan_demo()
print(f"Clústeres hallados: {resultado['n_clusters']}")
print(f"Puntos de ruido: {resultado['n_noise']}")
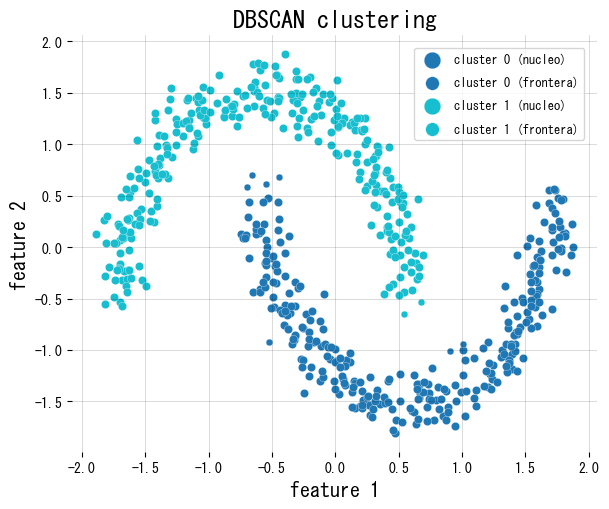
4. Consejos prácticos #
- Traza el gráfico de distancias ordenadas al k-ésimo vecino (k =
min_samples) y eligeepsdonde aparezca el codo. - Usa pipelines para estandarizar y agrupar en un mismo paso.
- Para conjuntos de datos grandes considera índices de vecinos aproximados o HDBSCAN, que extiende DBSCAN a múltiples densidades y reduce la sensibilidad a
eps.
5. Referencias #
- Ester, M., Kriegel, H.-P., Sander, J., & Xu, X. (1996). A Density-Based Algorithm for Discovering Clusters in Large Spatial Databases with Noise. KDD.
- Schubert, E., Sander, J., Ester, M., Kriegel, H.-P., & Xu, X. (2017). DBSCAN Revisited, Revisited. ACM Transactions on Database Systems.
- scikit-learn developers. (2024). Clustering. https://scikit-learn.org/stable/modules/clustering.html