2.5.3
X-means
Resumen
- X-means amplía K-means y estima automáticamente el número de clústeres mediante criterios BIC/MDL.
- Los parámetros de clúster mínimo y máximo acotan la búsqueda y evitan particiones excesivas.
- Comparar K-means con k fijo frente a X-means ayuda a medir el impacto de la selección de modelo en la interpretación.
Intuicion #
X-means aplica una estrategia de dividir y validar: comienza con pocos clústeres y solo divide cuando el criterio estadístico mejora. Así reduce la exploración manual de k sin perder control sobre la complejidad.
Explicacion Detallada #
X-means es un tipo de algoritmo de agrupamiento que determina automáticamente el número de clústeres a medida que avanza el proceso de agrupamiento. Esta página compara los resultados de k-means++ y X-means.
Pelleg, Dan, y Andrew W. Moore. “X-means: Extending k-means with efficient estimation of the number of clusters.” Icml. Vol. 1. 2000.
import numpy as np
import matplotlib.pyplot as plt
from sklearn.cluster import KMeans
from sklearn.datasets import make_blobs
Cuando k está predefinido en k-means #
def plot_by_kmeans(X, k=5):
y_pred = KMeans(n_clusters=k, random_state=random_state, init="random").fit_predict(
X
)
plt.scatter(X[:, 0], X[:, 1], c=y_pred, marker="x")
plt.title(f"k-means, n_clusters={k}")
# Crear datos de muestra
n_samples = 1000
random_state = 117117
X, _ = make_blobs(
n_samples=n_samples, random_state=random_state, cluster_std=1, centers=10
)
# Ejecutar k-means++.
plot_by_kmeans(X)

Ejecutar sin especificar el número de clústeres en X-means #
BIC (criterio de información bayesiano) #
from pyclustering.cluster.xmeans import xmeans
from pyclustering.cluster.center_initializer import kmeans_plusplus_initializer
BAYESIAN_INFORMATION_CRITERION = 0
MINIMUM_NOISELESS_DESCRIPTION_LENGTH = 1
def plot_by_xmeans(
X, c_min=3, c_max=10, criterion=BAYESIAN_INFORMATION_CRITERION, tolerance=0.025
):
initial_centers = kmeans_plusplus_initializer(X, c_min).initialize()
xmeans_instance = xmeans(
X, initial_centers, c_max, criterion=criterion, tolerance=tolerance
)
xmeans_instance.process()
# Crear datos para las parcelas
clusters = xmeans_instance.get_clusters()
n_samples = X.shape[0]
c = []
for i, cluster_i in enumerate(clusters):
X_ci = X[cluster_i]
color_ci = [i for _ in cluster_i]
plt.scatter(X_ci[:, 0], X_ci[:, 1], marker="x")
plt.title("x-means")
# Ejecutar x-means
plot_by_xmeans(X, c_min=3, c_max=10, criterion=BAYESIAN_INFORMATION_CRITERION)
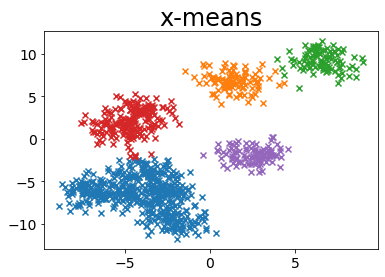
MINIMUM_NOISELESS_DESCRIPTION_LENGTH #
plot_by_xmeans(X, c_min=3, c_max=10, criterion=MINIMUM_NOISELESS_DESCRIPTION_LENGTH)
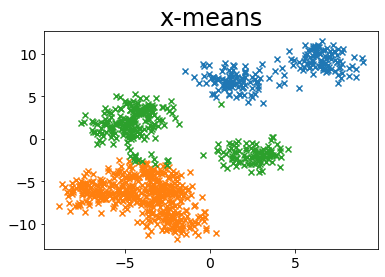
Influencia del parámetro de tolerancia #
X, _ = make_blobs(
n_samples=2000,
random_state=random_state,
cluster_std=0.4,
centers=10,
)
plt.figure(figsize=(25, 5))
for i, ti in enumerate(np.linspace(0.0001, 1, 5)):
ti = np.round(ti, 4)
plt.subplot(1, 10, i + 1)
plot_by_xmeans(
X, c_min=3, c_max=10, criterion=BAYESIAN_INFORMATION_CRITERION, tolerance=ti
)
plt.title(f"tol={ti}")

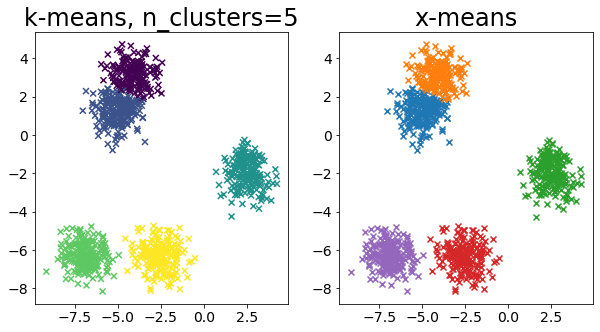
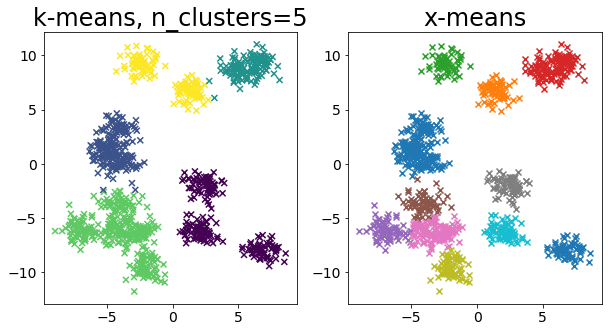
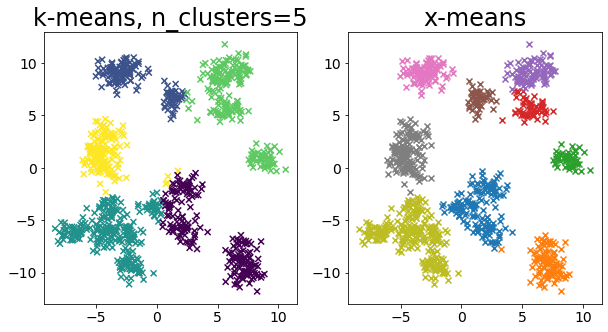
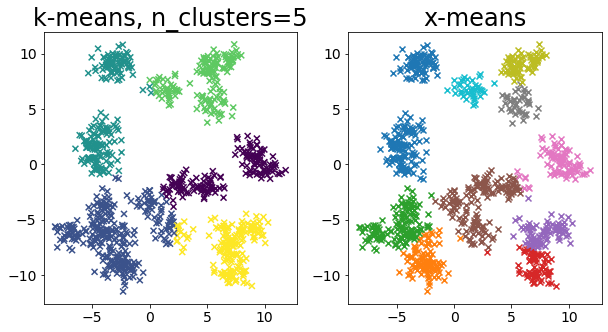
Comparar k-means y X-means para varios conjuntos de datos #
for i in range(5):
X, _ = make_blobs(
n_samples=n_samples,
random_state=random_state,
cluster_std=0.7,
centers=5 + i * 5,
)
plt.figure(figsize=(10, 5))
plt.subplot(1, 2, 1)
plot_by_kmeans(X)
plt.subplot(1, 2, 2)
plot_by_xmeans(X, c_min=3, c_max=20)
plt.show()