2.6.1
PCA
Resumen
- PCA encuentra ejes ortogonales de maxima varianza y proyecta los datos sobre ellos.
- La varianza explicada permite elegir cuantas componentes conservar.
- La estandarizacion es clave cuando las variables tienen escalas distintas.
Intuicion #
PCA rota el sistema de coordenadas hacia direcciones informativas. Conservando pocos ejes principales se reduce dimension sin perder la estructura dominante.
Explicacion Detallada #
import numpy as np
import matplotlib.pyplot as plt
import japanize_matplotlib
from sklearn.datasets import make_blobs
Datos para experimentos #
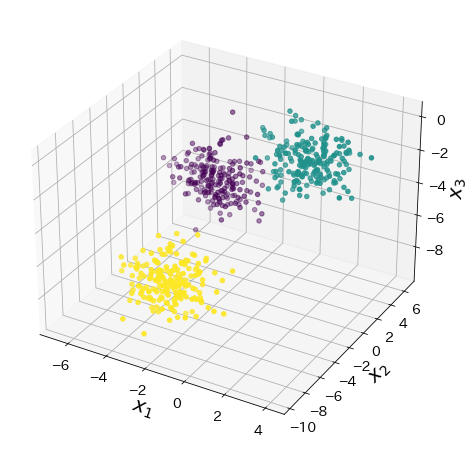
X, y = make_blobs(n_samples=600, n_features=3, random_state=117117)
fig = plt.figure(figsize=(8, 8))
ax = fig.add_subplot(projection="3d")
ax.scatter(X[:, 0], X[:, 1], X[:, 2], marker="o", c=y)
ax.set_xlabel("$x_1$")
ax.set_ylabel("$x_2$")
ax.set_zlabel("$x_3$")
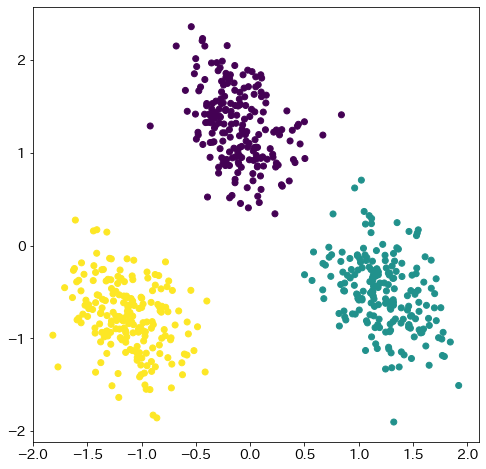
Reducir la dimensionalidad a dos dimensiones con PCA #
from sklearn.decomposition import PCA
from sklearn.preprocessing import StandardScaler
pca = PCA(n_components=2, whiten=True)
X_pca = pca.fit_transform(StandardScaler().fit_transform(X))
fig = plt.figure(figsize=(8, 8))
plt.scatter(X_pca[:, 0], X_pca[:, 1], c=y)
Observar el efecto de la normalización #
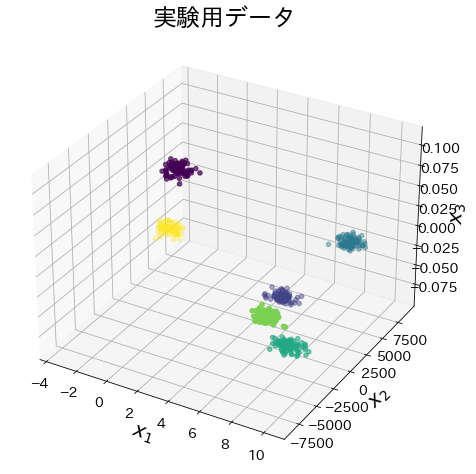
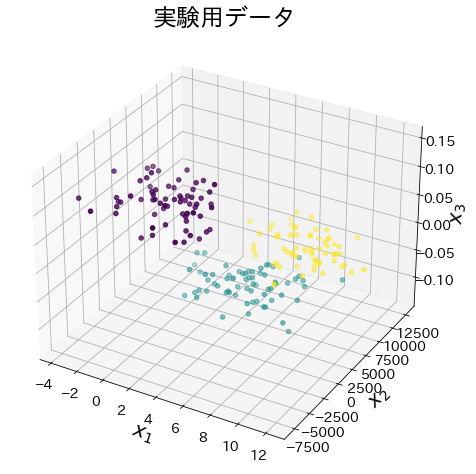
Número de clústeres: 3, con superposición entre clústeres #
# Datos experimentales
X, y = make_blobs(
n_samples=200, n_features=3, random_state=11711, centers=3, cluster_std=2.0
)
X[:, 1] = X[:, 1] * 1000
X[:, 2] = X[:, 2] * 0.01
X_ss = StandardScaler().fit_transform(X)
# Graficar los datos originales
fig = plt.figure(figsize=(8, 8))
ax = fig.add_subplot(projection="3d")
ax.scatter(X[:, 0], X[:, 1], X[:, 2], marker="o", c=y)
ax.set_xlabel("$x_1$")
ax.set_ylabel("$x_2$")
ax.set_zlabel("$x_3$")
plt.title("Datos experimentales")
plt.show()
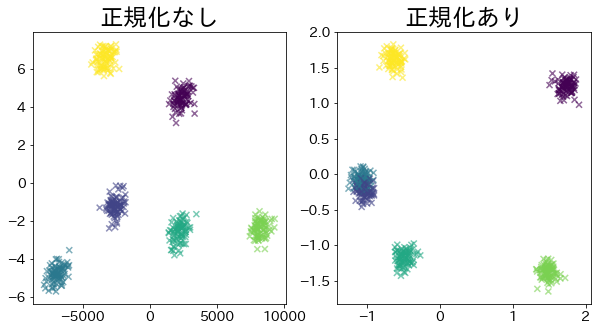
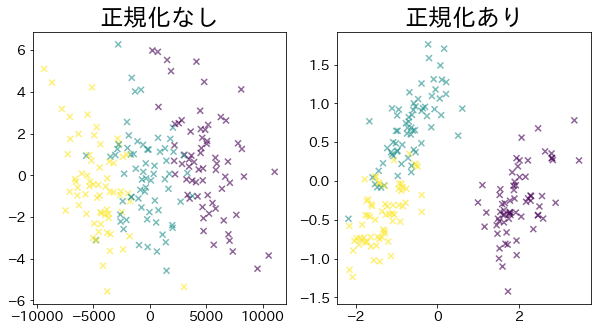
# PCA sin normalización
pca = PCA(n_components=2).fit(X)
X_pca = pca.transform(X)
# PCA con normalización
pca_ss = PCA(n_components=2).fit(X_ss)
X_pca_ss = pca_ss.transform(X_ss)
fig = plt.figure(figsize=(10, 5))
plt.subplot(121)
plt.title("Sin normalización")
plt.scatter(X_pca[:, 0], X_pca[:, 1], c=y, marker="x", alpha=0.6)
plt.subplot(122)
plt.title("Con normalización")
plt.scatter(X_pca_ss[:, 0], X_pca_ss[:, 1], c=y, marker="x", alpha=0.6)
Número de clústeres: 6, sin superposición entre clústeres #
# Datos experimentales
X, y = make_blobs(
n_samples=500, n_features=3, random_state=11711, centers=6, cluster_std=0.4
)
X[:, 1] = X[:, 1] * 1000
X[:, 2] = X[:, 2] * 0.01
X_ss = StandardScaler().fit_transform(X)
# Graficar los datos originales
fig = plt.figure(figsize=(8, 8))
ax = fig.add_subplot(projection="3d")
ax.scatter(X[:, 0], X[:, 1], X[:, 2], marker="o", c=y)
ax.set_xlabel("$x_1$")
ax.set_ylabel("$x_2$")
ax.set_zlabel("$x_3$")
plt.title("Datos experimentales")
plt.show()
# PCA sin normalización
pca = PCA(n_components=2).fit(X)
X_pca = pca.transform(X)
# PCA con normalización
pca_ss = PCA(n_components=2).fit(X_ss)
X_pca_ss = pca_ss.transform(X_ss)
fig = plt.figure(figsize=(10, 5))
plt.subplot(121)
plt.title("Sin normalización")
plt.scatter(X_pca[:, 0], X_pca[:, 1], c=y, marker="x", alpha=0.6)
plt.subplot(122)
plt.title("Con normalización")
plt.scatter(X_pca_ss[:, 0], X_pca_ss[:, 1], c=y, marker="x", alpha=0.6)