2.1.9
Regresión Partial Least Squares (PLS)
Resumen
- PLS extrae factores latentes que maximizan la covarianza entre predictores y objetivo antes de realizar la regresión.
- A diferencia de PCA, los ejes aprendidos incorporan información de la variable objetivo, manteniendo el desempeño predictivo mientras se reduce la dimensionalidad.
- Ajustar el número de factores latentes estabiliza el modelo cuando existe fuerte multicolinealidad.
- Analizar los loadings permite identificar qué combinaciones de características se relacionan más con la variable objetivo.
Intuicion #
Este metodo se entiende mejor al conectar sus supuestos con la estructura de los datos y su efecto en la generalizacion.
Explicacion Detallada #
Formulación matemática #
Dado un matriz de predictores \(\mathbf{X}\) y un vector objetivo \(\mathbf{y}\), PLS alterna las actualizaciones de las puntuaciones latentes \(\mathbf{t} = \mathbf{X} \mathbf{w}\) y \(\mathbf{u} = \mathbf{y} c\) de forma que la covarianza \(\mathbf{t}^\top \mathbf{u}\) sea máxima. Repetir el proceso produce un conjunto de factores latentes sobre el que se ajusta un modelo lineal
$$ \hat{y} = \mathbf{t} \boldsymbol{b} + b_0. $$El número de factores \(k\) suele elegirse mediante validación cruzada.
Experimentos con Python #
Comparamos el desempeño de PLS para distintos números de factores latentes con el conjunto de datos de acondicionamiento físico de Linnerud.
| |
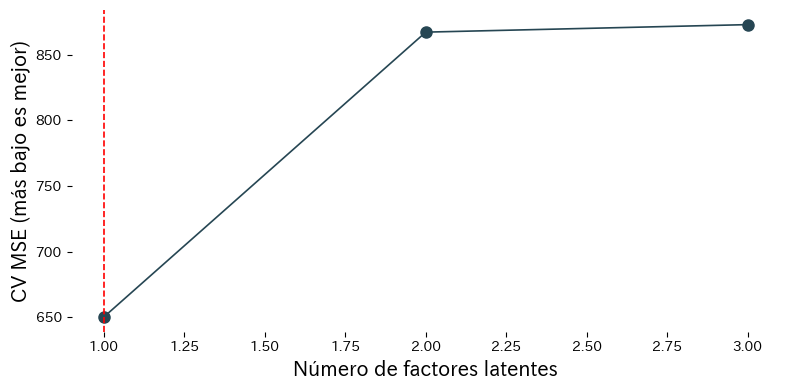
Interpretación de los resultados #
- El MSE de validación cruzada desciende al añadir factores, alcanza un mínimo y luego empeora si seguimos agregando más.
- Inspeccionar
x_loadings_yy_loadings_muestra qué características contribuyen más a cada factor latente. - Estandarizar las entradas garantiza que características con diferentes escalas aporten de manera equilibrada.
Referencias #
- Wold, H. (1975). Soft Modelling by Latent Variables: The Non-Linear Iterative Partial Least Squares (NIPALS) Approach. En Perspectives in Probability and Statistics. Academic Press.
- Geladi, P., & Kowalski, B. R. (1986). Partial Least-Squares Regression: A Tutorial. Analytica Chimica Acta, 185, 1–17.