2.1.8
Regresión por componentes principales (PCR)
Resumen
- PCR aplica PCA para comprimir las características antes de ajustar la regresión lineal, reduciendo la inestabilidad causada por la multicolinealidad.
- Los componentes principales priorizan las direcciones con mayor varianza, filtrando el ruido y preservando la estructura informativa.
- Elegir cuántos componentes conservar permite equilibrar el riesgo de sobreajuste y el coste computacional.
- Un buen preprocesamiento —estandarización y tratamiento de valores perdidos— sienta las bases para un modelo preciso e interpretable.
Intuicion #
Este metodo se entiende mejor al conectar sus supuestos con la estructura de los datos y su efecto en la generalizacion.
Explicacion Detallada #
Formulación matemática #
Aplicamos PCA a la matriz de diseño estandarizada \(\mathbf{X}\) y conservamos los \(k\) autovectores principales. Con las puntuaciones \(\mathbf{Z} = \mathbf{X} \mathbf{W}_k\), el modelo de regresión
$$ y = \boldsymbol{\gamma}^\top \mathbf{Z} + b $$se ajusta a los datos. Los coeficientes en el espacio original se recuperan mediante \(\boldsymbol{\beta} = \mathbf{W}_k \boldsymbol{\gamma}\). El número de componentes \(k\) se elige usando la varianza explicada acumulada o validación cruzada.
Experimentos con Python #
Evaluamos la regresión PCR en el conjunto de datos de diabetes variando el número de componentes y midiendo el desempeño con validación cruzada.
from __future__ import annotations
import japanize_matplotlib
import matplotlib.pyplot as plt
import numpy as np
from sklearn.datasets import load_diabetes
from sklearn.decomposition import PCA
from sklearn.linear_model import LinearRegression
from sklearn.model_selection import cross_val_score
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
def evaluate_pcr_components(
cv_folds: int = 5,
xlabel: str = "Number of components k",
ylabel: str = "CV MSE (lower is better)",
title: str | None = None,
label_best: str = "best={k}",
) -> dict[str, float]:
"""Cross-validate PCR with varying component counts and plot the curve.
Args:
cv_folds: Number of folds for cross-validation.
xlabel: Label for the component-count axis.
ylabel: Label for the error axis.
title: Optional title for the plot.
label_best: Format string for highlighting the best component count.
Returns:
Dictionary containing the best component count and its CV score.
"""
japanize_matplotlib.japanize()
X, y = load_diabetes(return_X_y=True)
def build_pcr(n_components: int) -> Pipeline:
return Pipeline([
("scale", StandardScaler()),
("pca", PCA(n_components=n_components, random_state=0)),
("reg", LinearRegression()),
])
components = np.arange(1, X.shape[1] + 1)
cv_scores = []
for k in components:
model = build_pcr(int(k))
score = cross_val_score(
model,
X,
y,
cv=cv_folds,
scoring="neg_mean_squared_error",
)
cv_scores.append(score.mean())
cv_scores_arr = np.array(cv_scores)
best_idx = int(np.argmax(cv_scores_arr))
best_k = int(components[best_idx])
best_mse = float(-cv_scores_arr[best_idx])
best_model = build_pcr(best_k).fit(X, y)
explained = best_model["pca"].explained_variance_ratio_
fig, ax = plt.subplots(figsize=(8, 4))
ax.plot(components, -cv_scores_arr, marker="o")
ax.axvline(best_k, color="red", linestyle="--", label=label_best.format(k=best_k))
ax.set_xlabel(xlabel)
ax.set_ylabel(ylabel)
if title:
ax.set_title(title)
ax.legend()
fig.tight_layout()
plt.show()
return {
"best_k": best_k,
"best_mse": best_mse,
"explained_variance_ratio": explained,
}
metrics = evaluate_pcr_components(
xlabel="Número de componentes k",
ylabel="MSE de CV (más bajo es mejor)",
title="PCR y número de componentes",
label_best="mejor k={k}",
)
print(f"Mejor número de componentes: {metrics['best_k']}")
print(f"Mejor MSE de CV: {metrics['best_mse']:.3f}")
print("Proporción de varianza explicada:", metrics['explained_variance_ratio'])
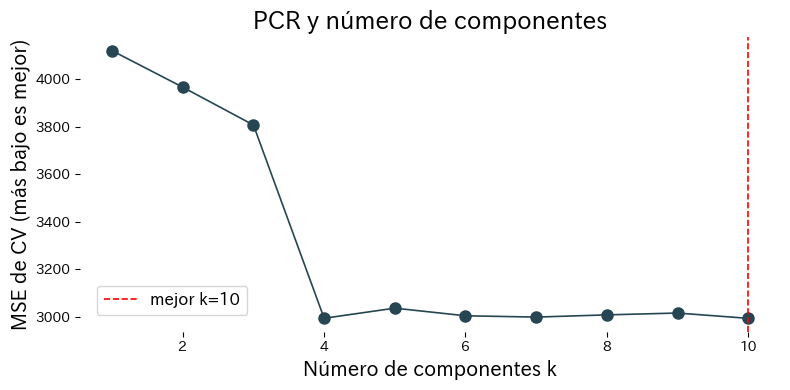
Interpretación de los resultados #
- Al aumentar los componentes, el ajuste sobre el entrenamiento mejora, pero el MSE de validación cruzada alcanza un mínimo intermedio.
- La proporción de varianza explicada indica cuánta variabilidad captura cada componente.
- Analizar los loadings muestra qué características originales contribuyen más a cada dirección principal.
Referencias #
- Jolliffe, I. T. (2002). Principal Component Analysis (2nd ed.). Springer.
- Massy, W. F. (1965). Principal Components Regression in Exploratory Statistical Research. Journal of the American Statistical Association, 60(309), 234–256.