2.3.3
Parámetros del árbol
- Los árboles de decisión ofrecen varios mandos—profundidad, muestras mínimas por división/hoja, poda y pesos de clase—que determinan directamente su capacidad e interpretabilidad.
max_depthymin_samples_leaflimitan el detalle de las reglas, mientras queccp_alpha(poda por complejidad de costo) elimina ramas cuyo aporte no justifica su tamaño.- Escoger el criterio adecuado (
squared_error,absolute_error,friedman_mse, etc.) cambia la sensibilidad del árbol frente a atípicos. - Las visualizaciones de fronteras de decisión y de la estructura del árbol facilitan explicar por qué una determinada combinación de hiperparámetros funciona.
Intuicion #
Este metodo se entiende mejor al conectar sus supuestos con la estructura de los datos y su efecto en la generalizacion.
Explicacion Detallada #
1. Descripción general #
Sin restricciones, un árbol sigue dividiendo hasta dejar hojas totalmente puras, lo que suele implicar sobreajuste. Los hiperparámetros actúan como regularizadores: la profundidad impide que el árbol sea demasiado detallado, las muestras mínimas evitan hojas minúsculas y la poda colapsa ramas marginales.
2. Ganancia de impureza y poda por complejidad #
Para un nodo padre (P) que se divide en hijos (L) y (R), la reducción de impureza es
$$ \Delta I = I(P) - \frac{|L|}{|P|} I(L) - \frac{|R|}{|P|} I(R), $$donde (I(\cdot)) puede ser Gini, entropía, MSE o MAE. Sólo mantenemos divisiones con (\Delta I > 0).
La poda por complejidad evalúa un árbol (T) mediante
$$ R_\alpha(T) = R(T) + \alpha |T|, $$donde (R(T)) es la pérdida de entrenamiento, (|T|) el número de hojas y (\alpha \ge 0) penaliza estructuras grandes. Aumentar (\alpha) conduce a árboles más simples.
3. Experimentos en Python #
El siguiente código entrena varios DecisionTreeRegressor sobre un conjunto sintético y muestra cómo distintos parámetros afectan el (R^2) de entrenamiento y prueba.
| |
Las siguientes figuras muestran cómo cambia la superficie de predicción al modificar los parámetros clave:
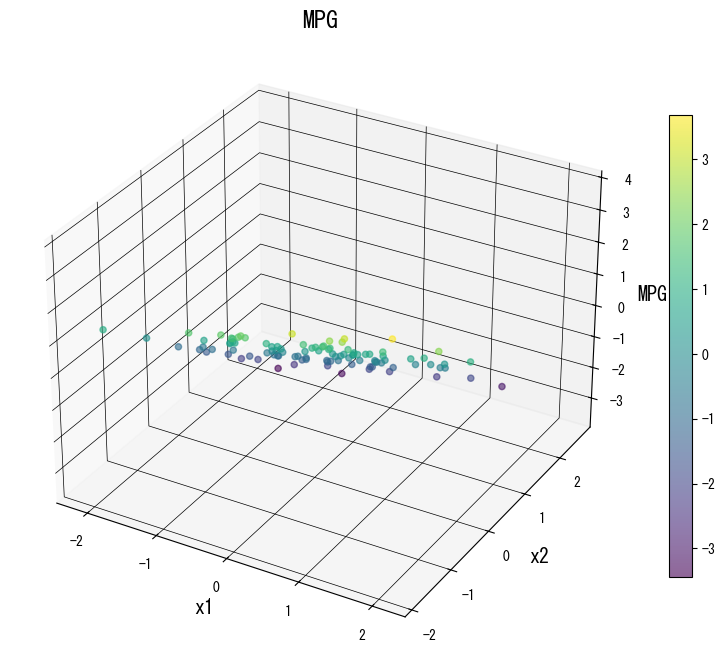
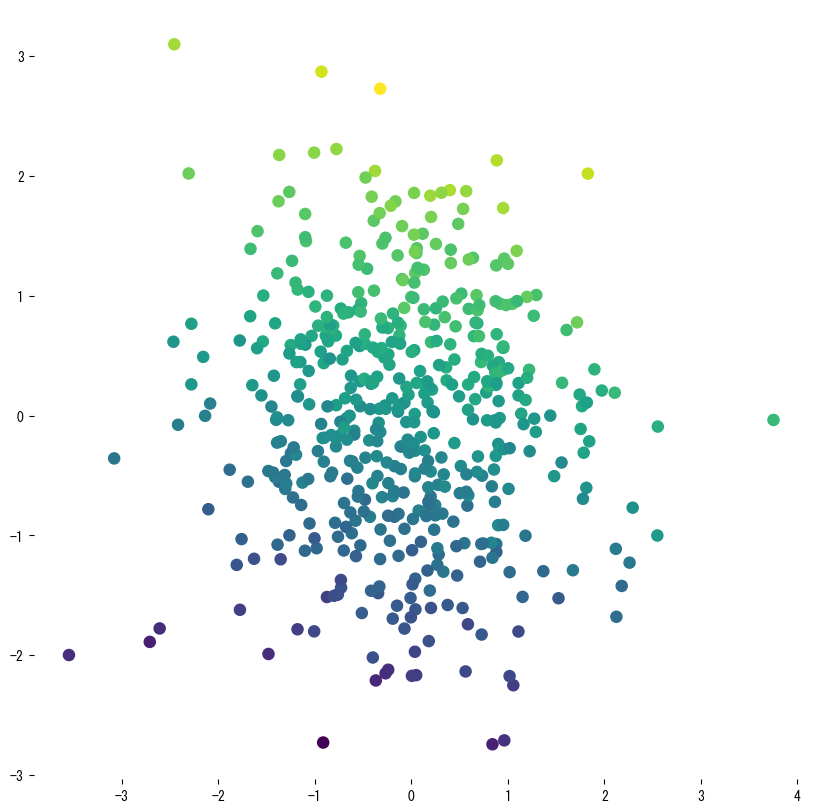
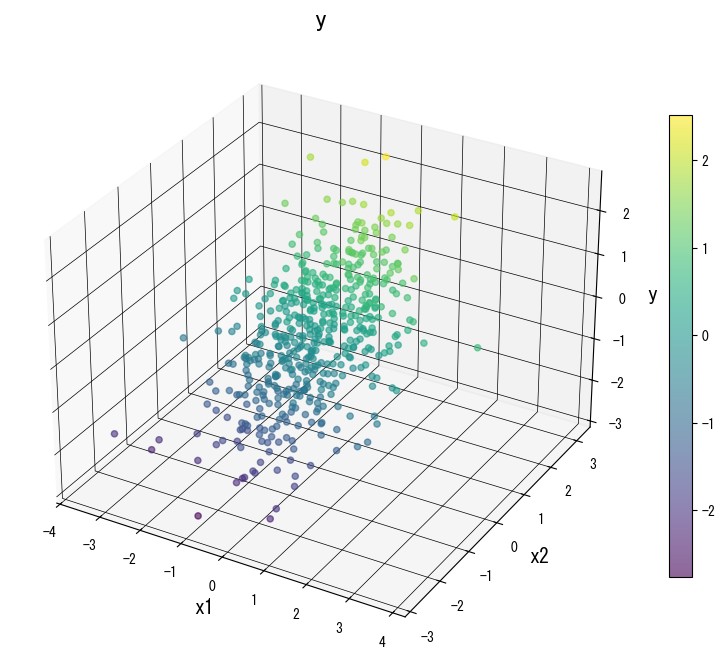
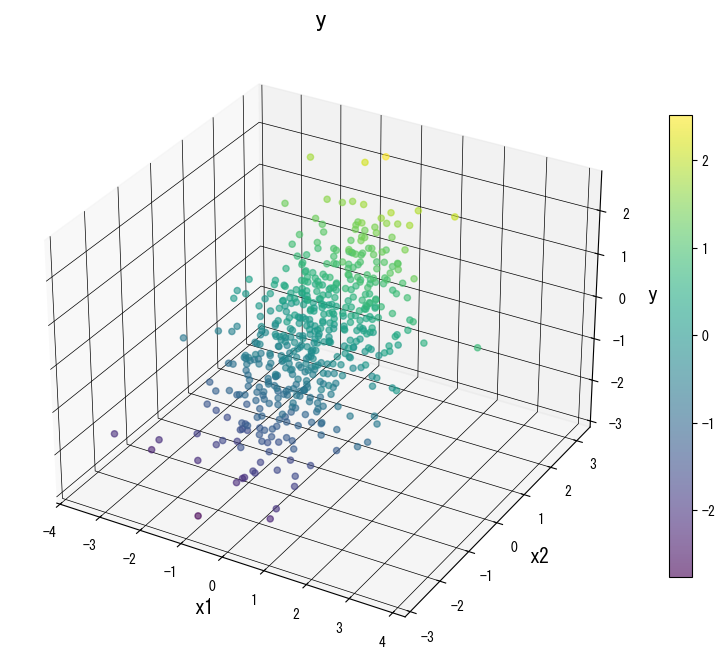
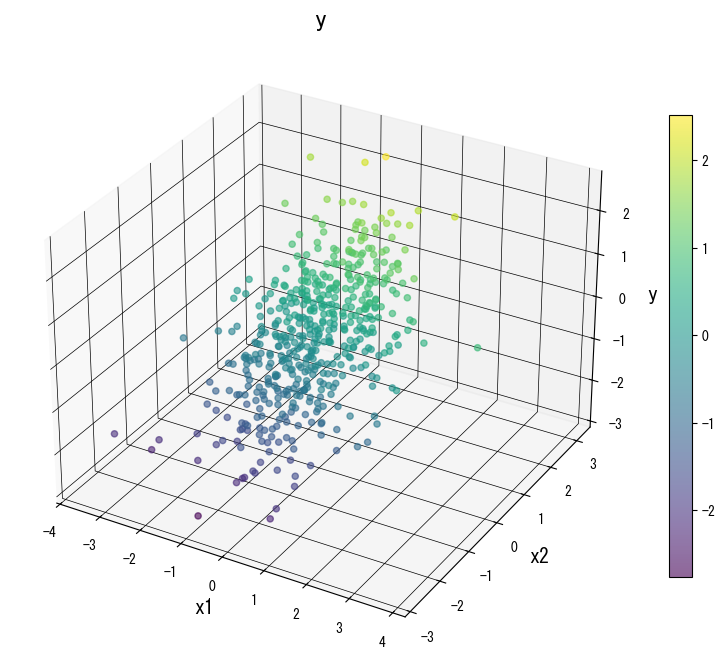
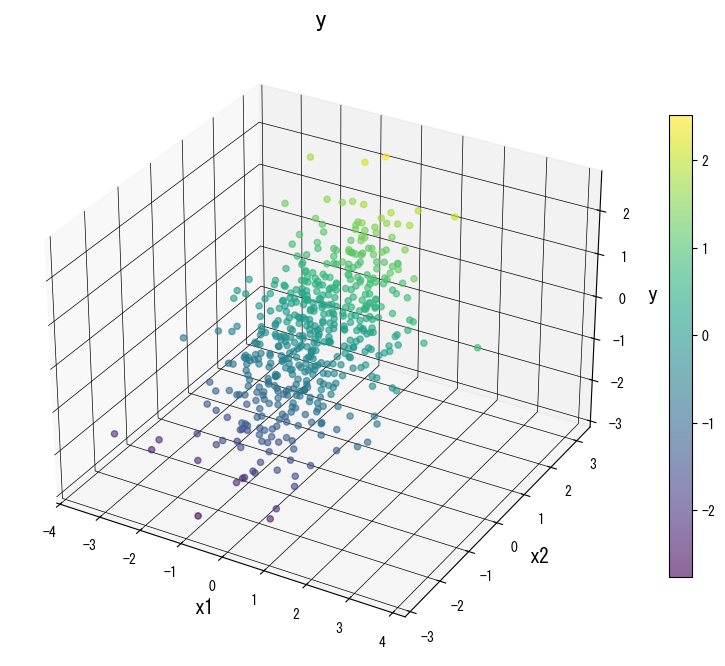
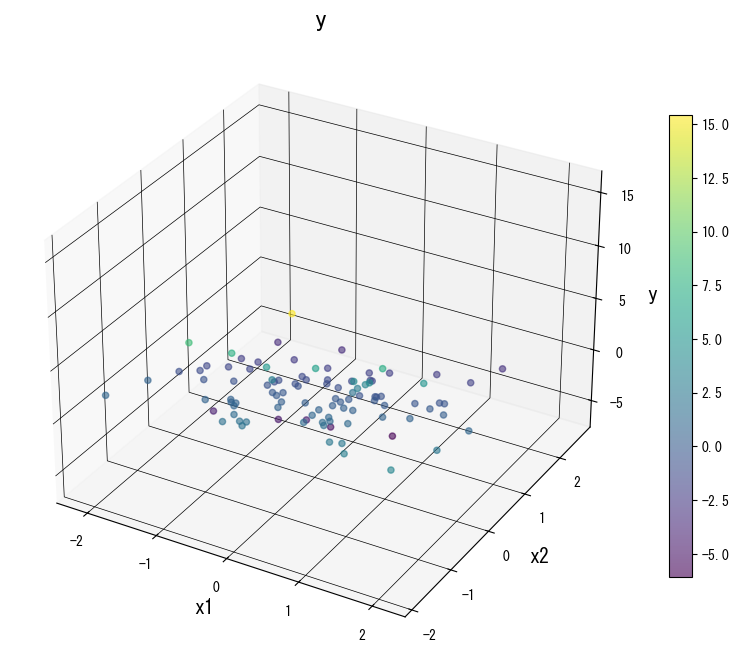
4. Referencias #
- Breiman, L., Friedman, J. H., Olshen, R. A., & Stone, C. J. (1984). Classification and Regression Trees. Wadsworth.
- Breiman, L., & Friedman, J. H. (1991). Cost-Complexity Pruning. En Classification and Regression Trees. Chapman & Hall.
- scikit-learn developers. (2024). Decision Trees. https://scikit-learn.org/stable/modules/tree.html