2.5.4
DBSCAN
- DBSCAN mengelompokkan titik berdasarkan kepadatan lokal sehingga bentuk klaster bisa bebas dan area jarang otomatis jadi noise.
- Dua hiperparameter utama:
eps(radius tetangga) danmin_samples(jumlah minimum tetangga untuk menjadi titik inti). - Setiap titik diklasifikasikan sebagai inti, tepi, atau noise; klaster terbentuk dari komponen terhubung titik inti beserta tetangga tepinya.
- Biasanya
min_samplesdipatok terlebih dahulu (≥ dimensi + 1) laluepsdisapu sambil memonitor porsi titik yang berubah menjadi noise.
Intuisi #
Metode ini dipahami lewat asumsi dasarnya, karakteristik data, dan dampak pengaturan parameter terhadap generalisasi.
Penjelasan Rinci #
1. Gambaran umum #
DBSCAN tidak memerlukan jumlah klaster di muka. Ia menginspeksi tiap sampel:
- Inti: punya ≥
min_samplestetangga dalam jarakeps. - Tepi: berada dalam radius
epsdari titik inti tetapi tidak memenuhi syarat inti sendiri. - Noise: tidak berada pada tetangga inti mana pun.
Pendekatan ini membuat DBSCAN tangguh untuk pola berbentuk bulan sabit atau cincin. Pastikan fitur sudah diskalakan agar jarak bermakna.
2. Definisi formal #
Untuk (x_i \in \mathcal{X}), tetangga (\varepsilon)-nya adalah
$$ \mathcal{N}_\varepsilon(x_i) = \{ x_j \in \mathcal{X} \mid \lVert x_i - x_j \rVert \le \varepsilon \}. $$Jika (|\mathcal{N}_\varepsilon(x_i)| \ge \texttt{min_samples}|) maka titik inti. DBSCAN memperluas klaster dengan menjelajah titik yang dapat dijangkau secara densitas dan menandai sisanya sebagai noise. Kompleksitasnya (O(n \log n)) bila memakai indeks spasial.
3. Contoh Python #
Contoh berikut menjalankan DBSCAN pada dataset dua bulan sabit, membedakan warna inti/tepi, dan menghitung jumlah noise.
| |
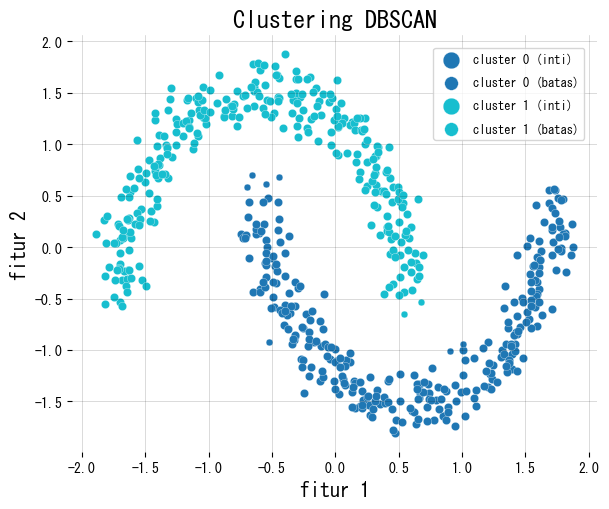
4. Tips praktis #
- Gunakan grafik jarak tetangga ke-k (k =
min_samples) untuk menemukanepsdi titik siku. - Jalankan penyeragaman fitur dan clustering di pipeline yang sama.
- Untuk data besar, pertimbangkan pendekatan HDBSCAN atau struktur tetangga mendekati agar pencarian tetangga lebih hemat.
5. Referensi #
- Ester, M., Kriegel, H.-P., Sander, J., & Xu, X. (1996). A Density-Based Algorithm for Discovering Clusters in Large Spatial Databases with Noise. KDD.
- Schubert, E., Sander, J., Ester, M., Kriegel, H.-P., & Xu, X. (2017). DBSCAN Revisited, Revisited. ACM Transactions on Database Systems.
- scikit-learn developers. (2024). Clustering. https://scikit-learn.org/stable/modules/clustering.html