2.1.8
Regresi Komponen Utama (PCR)
Ringkasan
- PCR menerapkan PCA untuk memampatkan fitur sebelum menjalankan regresi linear, sehingga mengurangi ketidakstabilan akibat multikolinearitas.
- Komponen utama memprioritaskan arah dengan varians besar, menyaring sumbu yang didominasi noise sambil mempertahankan struktur informatif.
- Menentukan jumlah komponen yang disimpan menyeimbangkan risiko overfitting dan biaya komputasi.
- Praproses yang baik—standarisasi dan penanganan nilai hilang—menjadi fondasi akurasi dan interpretabilitas.
Intuisi #
Metode ini dipahami lewat asumsi dasarnya, karakteristik data, dan dampak pengaturan parameter terhadap generalisasi.
Penjelasan Rinci #
Formulasi matematis #
PCA diterapkan pada matriks rancangan \(\mathbf{X}\) yang telah distandarkan, lalu \(k\) autovektor teratas dipertahankan. Dengan skor komponen utama \(\mathbf{Z} = \mathbf{X} \mathbf{W}_k\), model regresi
$$ y = \boldsymbol{\gamma}^\top \mathbf{Z} + b $$dipelajari. Koefisien dalam ruang fitur asli dipulihkan melalui \(\boldsymbol{\beta} = \mathbf{W}_k \boldsymbol{\gamma}\). Jumlah komponen \(k\) dipilih menggunakan varian kumulatif atau cross-validation.
Eksperimen dengan Python #
Kami menghitung skor validasi silang PCR pada dataset diabetes sambil memvariasikan jumlah komponen.
| |
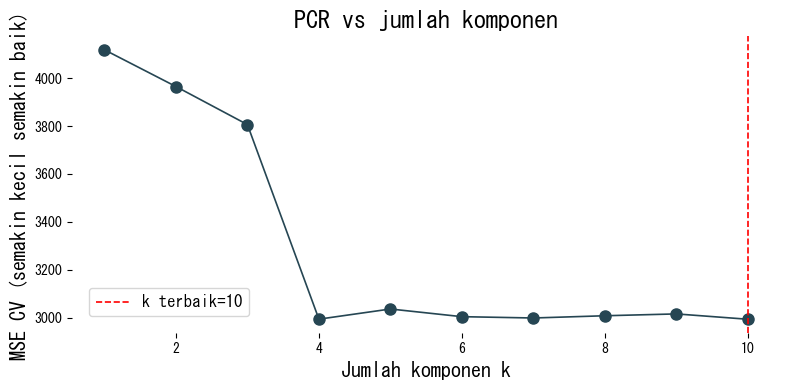
Cara membaca hasil #
- Semakin banyak komponen, kecocokan terhadap data latih meningkat, tetapi MSE validasi silang mencapai minimum pada jumlah menengah.
- Rasio varians yang dijelaskan memperlihatkan seberapa besar variabilitas yang ditangkap tiap komponen.
- Loading komponen menunjukkan fitur asli mana yang paling berkontribusi pada setiap arah utama.
Referensi #
- Jolliffe, I. T. (2002). Principal Component Analysis (2nd ed.). Springer.
- Massy, W. F. (1965). Principal Components Regression in Exploratory Statistical Research. Journal of the American Statistical Association, 60(309), 234–256.