3.3.4
RobustScaler
まとめ
- メディアンと四分位範囲(IQR)を用いて、外れ値に頑健なスケーリングを行う。
sklearn.preprocessing.RobustScalerのfit_transformで変換し、quantile_rangeで IQR の範囲を調整する。- 極端な外れ値が含まれるデータで、スケールが歪まない正規化を行いたいときに使う。
- 標準化(StandardScaler)や Min-Max 正規化の概念を先に学ぶと理解がスムーズです
<p><b>RobustScaler</b> はメディアン(第 50 パーセンタイル)と四分位範囲 \(IQR = Q_3 - Q_1\) を用いてスケーリングします。極端な外れ値が存在してもスケールが大きく歪まない点が特徴です。</p>
1. 定義 #
観測値 (x)、メディアン (m = \operatorname{median}(x))、IQR に対し、
$$ z = \frac{x - m}{\operatorname{IQR}} $$と変換します。quantile_range=(q_1, q_3) を変更すると IQR の定義域を調整できます。
2. Pythonで計算 #
| |
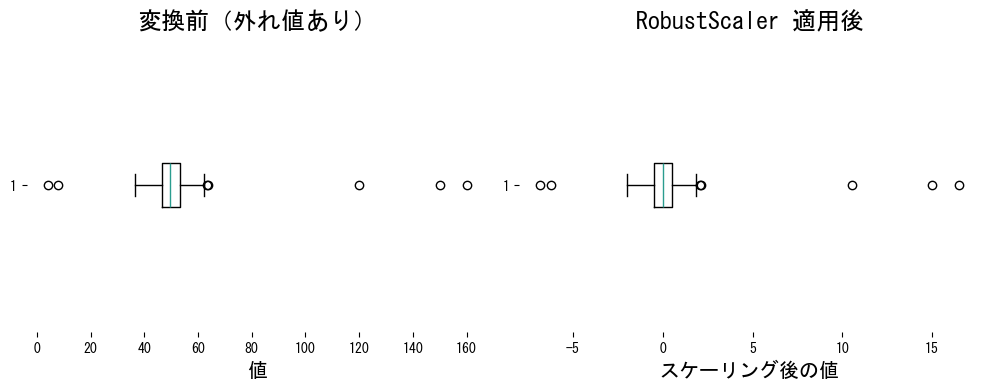
3. 他のスケーラーとの比較 #
| スケーラー | 中心化 | スケール基準 | 外れ値耐性 |
|---|---|---|---|
| StandardScaler | 平均 | 標準偏差 | 低い |
| MinMaxScaler | 最小値 | max − min | 低い |
| RobustScaler | メディアン | IQR | 高い |
外れ値が多いデータではStandardScalerの平均と標準偏差が歪みやすいため、RobustScalerが第一候補になります。
4. quantile_rangeの調整 #
デフォルトは(25, 75)ですが、範囲を広げると外れ値の影響を受けやすく、狭めるとデータの中心付近だけでスケーリングします。
| |
一般に(25, 75)で十分ですが、外れ値が極端に多い場合は(10, 90)を試すとよいでしょう。
5. 実務でのポイント #
quantile_rangeを狭めると外れ値の影響をさらに抑えられますが、極端に狭い範囲では情報が失われやすいので注意してください。with_centering=Falseを指定すると中心化せずにスケーリングのみ行えます。疎行列でメディアンを引くとスパース性が失われるケースで有効です。- 学習データで
fitした scaler を推論時にtransformするのが基本です。デプロイ時には学習済み scaler を必ず一緒に保存します。 - RobustScaler の後にさらに StandardScaler を重ねる必要は通常ありません。データ分布とモデル特性に合わせ、どのスケーリングが適切かを選択しましょう。
まとめ #
- RobustScalerはメディアンとIQRを用いたスケーリングで、外れ値に強い。
- StandardScalerやMinMaxScalerで結果が歪む場合の代替手段として有効。
- 疎行列では
with_centering=Falseを指定してスパース性を保つ。
- StandardScaler — 平均と標準偏差によるスケーリング
- MinMaxScaler — 最小値・最大値によるスケーリング