2.5.3
X-means
สรุป
- X-means เป็นส่วนขยายของ K-means ที่ประเมินการแยกคลัสเตอร์ด้วยเกณฑ์ BIC/MDL เพื่อประมาณจำนวนคลัสเตอร์อัตโนมัติ.
- การกำหนดจำนวนคลัสเตอร์ขั้นต่ำและสูงสุดช่วยจำกัดช่วงค้นหาและลดปัญหาแบ่งคลัสเตอร์มากเกินไป.
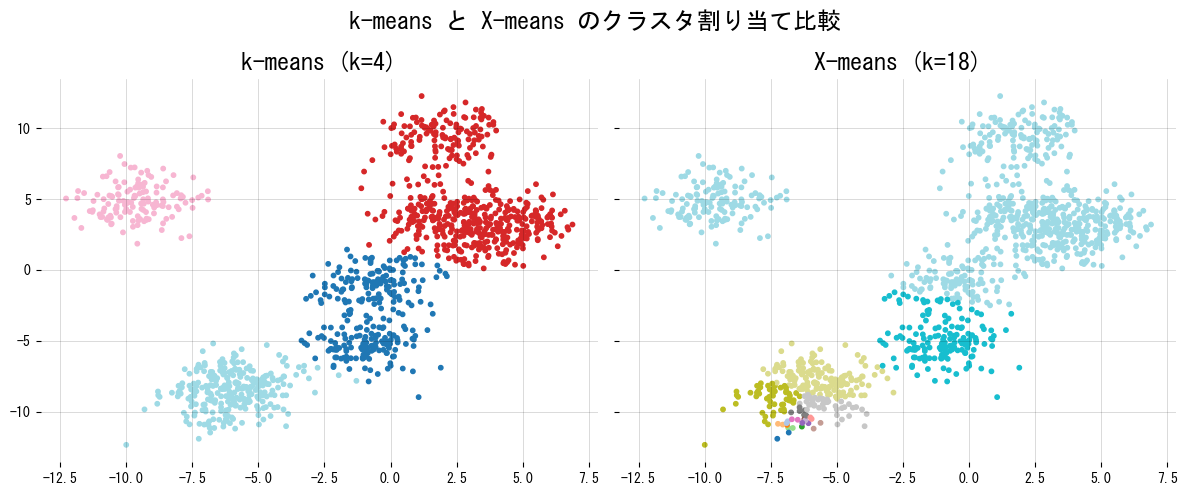
- การเปรียบเทียบกับ K-means แบบกำหนด k คงที่ช่วยเห็นผลของการเลือกจำนวนคลัสเตอร์ต่อการตีความข้อมูล.
สัญชาตญาณ #
X-means ใช้แนวคิดแบ่งแล้วทดสอบ เริ่มจากคลัสเตอร์หยาบก่อน แล้วจะแบ่งเพิ่มเฉพาะกรณีที่เกณฑ์สถิติชี้ว่าดีขึ้น วิธีนี้ลดการเดา k ด้วยมือและยังควบคุมความซับซ้อนได้ดี.
คำอธิบายโดยละเอียด #
X-means | ประมาณจำนวนคลัสเตอร์อัตโนมัติ #
สรุป
- X-means ขยาย k-means ให้เลือกจำนวนคลัสเตอร์เองโดยลองแบ่งคลัสเตอร์ย่อยตามความจำเป็น
- เมื่อแบ่งคลัสเตอร์หนึ่งเป็นสอง จะคำนวณเกณฑ์ BIC/MDL หากค่าดีขึ้นจึงยอมรับการแบ่ง
- สามารถเขียนเวอร์ชันง่ายๆ ได้ด้วยการรัน k-means และคำนวณ BIC ด้วยตัวเอง ไม่จำเป็นต้องมีไลบรารีเฉพาะ
- เหมาะกับสถานการณ์ที่กำหนด \(k\) ล่วงหน้าได้ยาก เพราะปล่อยให้ข้อมูลชี้นำจำนวนกลุ่มที่เหมาะสม
ภาพรวมเชิงสัญชาติญาณ #
k-means ต้องระบุ \(k\) ล่วงหน้า X-means แก้ด้วยการเริ่มจาก \(k\) เล็กๆ แล้วตรวจทุกคลัสเตอร์ว่าสมควรแบ่งต่อหรือไม่: ถ้าแบ่งแล้ว BIC ดีขึ้นก็เก็บทั้งสองคลัสเตอร์ใหม่ไว้พิจารณาต่อ เมื่อไม่มีคลัสเตอร์ไหนควรแบ่ง ตัวเลข \(k\) ก็ถูกกำหนดอัตโนมัติ
สูตรสำคัญ #
ให้คลัสเตอร์ \({C_1,\ldots,C_k}\) กับศูนย์กลาง \({\mu_j}\) BIC สำหรับโมเดลหนึ่งคำนวณได้จาก
$$ \mathrm{BIC} = \ln L - \tfrac{p}{2} \ln n, $$โดย \(L\) คือ likelihood, \(p\) จำนวนพารามิเตอร์ และ \(n\) จำนวนตัวอย่าง ถ้า BIC หลังแบ่งใหญ่กว่าแสดงว่าโมเดลใหม่ดีกว่า เราจึงยอมรับการแบ่งนั้น
ทดลองด้วย Python #
โค้ดต่อไปนี้สร้างเวอร์ชันอย่างง่ายของ X-means และเปรียบเทียบกับ k-means ที่ตั้ง \(k=4\) คงที่
from __future__ import annotations
import matplotlib.pyplot as plt
import numpy as np
from numpy.typing import NDArray
from sklearn.cluster import KMeans
from sklearn.datasets import make_blobs
def generate_dataset(
n_samples: int = 1200,
n_centers: int = 9,
cluster_std: float = 1.1,
random_state: int = 1707,
) -> NDArray[np.float64]:
features, _ = make_blobs(
n_samples=n_samples,
centers=n_centers,
cluster_std=cluster_std,
random_state=random_state,
)
return features
def compute_bic(
data: NDArray[np.float64],
labels: NDArray[np.int64],
centers: NDArray[np.float64],
) -> float:
"""คำนวณ BIC สำหรับผลลัพธ์ของ k-means"""
n_samples, n_features = data.shape
n_clusters = centers.shape[0]
cluster_sizes = np.bincount(labels, minlength=n_clusters)
sse = 0.0
for idx, center in enumerate(centers):
if cluster_sizes[idx] == 0:
continue
diffs = data[labels == idx] - center
sse += float((diffs**2).sum())
variance = sse / max(n_samples - n_clusters, 1)
variance = max(variance, 1e-6)
log_likelihood = 0.0
norm_const = -0.5 * n_features * np.log(2 * np.pi * variance)
for size in cluster_sizes:
if size > 0:
log_likelihood += size * norm_const - 0.5 * (size - 1)
n_params = n_clusters * (n_features + 1)
bic = log_likelihood - 0.5 * n_params * np.log(n_samples)
return float(bic)
def xmeans_split(
data: NDArray[np.float64],
k_max: int = 20,
random_state: int = 42,
) -> NDArray[np.int64]:
"""X-means อย่างง่าย: แยกคลัสเตอร์เมื่อ BIC ดีขึ้น"""
rng = np.random.default_rng(random_state)
pending = [np.arange(len(data))]
accepted: list[NDArray[np.int64]] = []
while pending:
indices = pending.pop()
subset = data[indices]
if len(indices) <= 2:
accepted.append(indices)
continue
parent_labels = np.zeros(len(indices), dtype=int)
parent_center = subset.mean(axis=0, keepdims=True)
bic_parent = compute_bic(subset, parent_labels, parent_center)
split_model = KMeans(
n_clusters=2,
n_init=10,
max_iter=200,
random_state=rng.integers(0, 1_000_000),
).fit(subset)
bic_children = compute_bic(subset, split_model.labels_, split_model.cluster_centers_)
if bic_children > bic_parent and (len(accepted) + len(pending) + 1) < k_max:
pending.append(indices[split_model.labels_ == 0])
pending.append(indices[split_model.labels_ == 1])
else:
accepted.append(indices)
labels = np.empty(len(data), dtype=int)
for cluster_id, cluster_indices in enumerate(accepted):
labels[cluster_indices] = cluster_id
return labels
def run_xmeans_demo(
random_state: int = 2024,
initial_k: int = 4,
k_max: int = 18,
) -> dict[str, object]:
"""เปรียบเทียบ k-means ธรรมดากับ X-means"""
data = generate_dataset(random_state=random_state)
kmeans_labels = KMeans(
n_clusters=initial_k,
n_init=10,
random_state=random_state,
).fit_predict(data)
xmeans_labels = xmeans_split(data, k_max=k_max, random_state=random_state + 99)
unique_xmeans = np.unique(xmeans_labels)
fig, axes = plt.subplots(1, 2, figsize=(12, 5), sharex=True, sharey=True)
axes[0].scatter(data[:, 0], data[:, 1], c=kmeans_labels, cmap="tab20", s=10)
axes[0].set_title(f"k-means (k={initial_k})")
axes[0].grid(alpha=0.2)
axes[1].scatter(data[:, 0], data[:, 1], c=xmeans_labels, cmap="tab20", s=10)
axes[1].set_title(f"X-means (k={len(unique_xmeans)})")
axes[1].grid(alpha=0.2)
fig.suptitle("เปรียบเทียบผลการจัดกลุ่มของ k-means และ X-means")
fig.tight_layout()
plt.show()
return {
"kmeans_clusters": int(initial_k),
"xmeans_clusters": int(len(unique_xmeans)),
"cluster_sizes": np.bincount(xmeans_labels).tolist(),
}
results = run_xmeans_demo()
print(f"k-means ใช้ k = {results['kmeans_clusters']}")
print(f"X-means ประมาณ k = {results['xmeans_clusters']}")
print(f"ขนาดคลัสเตอร์ของ X-means: {results['cluster_sizes']}")
เอกสารอ้างอิง #
- Pelleg, D., & Moore, A. W. (2000). X-means: Extending k-means with Efficient Estimation of the Number of Clusters. ICML.
- Bahmani, B., Moseley, B., Vattani, A., Kumar, R., & Vassilvitskii, S. (2012). Scalable k-means++. VLDB.
- scikit-learn developers. (2024). Clustering. https://scikit-learn.org/stable/modules/clustering.html